En otra entrada vimos el éxito de 'genoma' desde el punto de vista lingüístico. Ahora vamos a abordar otro éxito completamente diferente.
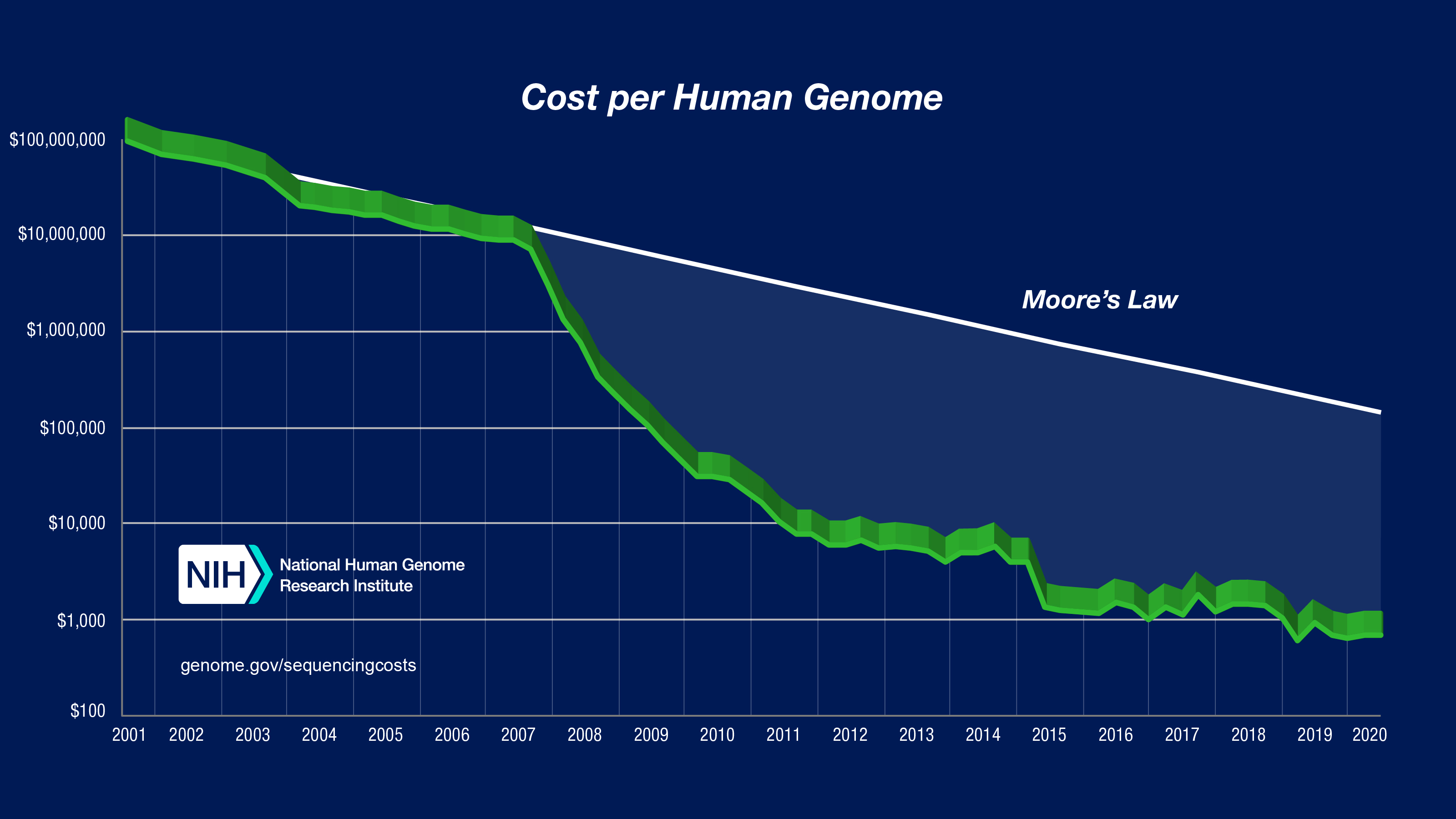
Cuando en 1984 se empezaron a plantear si sería posible secuenciar el genoma humano, el grupo de Craig Venter ya estaba secuenciando el primer procariota (Hemophilus imfluenzae) con el que dio a conocer el sistema de secuenciación indiscriminada (shotgun sequencing), y en Europa estábamos (sí, yo puse mi grano de arena) poniendo en marcha la secuenciación del primer eucariota (la levadura Saccharomyces cerevisiae). El debate estaba en que abordar la secuenciación de nuestro genoma iba a tener un coste excesivo dadas las pocas secuencias codificantes que contenía (que se creía, inocentemente, que constituían el 10%; si llegan a saber que solo era un 1,1%, igual ni empiezan). En la imagen adjunta se puede ver lo que costó el primer genoma humano y cómo ha ido decayendo el coste con los años y los avances tecnológicos.
Tras poner en canción a un montón de grupos e investigadores, en 2001 se publicó el primer borrador y se indicó que era 25 veces más grande que cualquier otro conocido por entonces. En 2003 se publicó la secuencia de la eucromatina humana (el 92,1%). Hoy, 20 años después del primer hito, sabemos que contiene 20 437 genes que codifican proteínas (coding genes) y otros 23 988 genes no codificantes (non-coding genes), en un genoma de 3,096 Gpb que produce más de 235.000 transcritos diferentes.
Se han secuenciado cientos de genomas, desde virus a eucariotas complejos, y se ha visto que el de muchos animales y plantas oscila entre 0,4 y 0,7 Gpb (un gigapar de bases corresponde a 1 millón de pares de bases). Por tanto, nuestro genoma ya no es más que unas 10 veces mayor que el de muchos otros seres vivos. Así que de 25× en 2001 hemos pasado a un escaso 10× en 2021. O lo que es más denigrante para tantísimos homocéntricos: nuestro genoma es solo ligeramente superior al del ratón (2,72 Gpb) y tiene menos genes codificantes que una mala hierba, el arroz y, en general, que cualquier planta. Así que ¿tendremos el genoma más grande del planeta? La respuesta llegará en la próxima nanoentrada.