El nanoblog del Gonz
Aquí desparramo lo que dan de sí mis sinapsis sobre ciencia, traducciones varias y todo lo demás
About.me/mgclaros
17 abr 2022
Los Cavendish y el DNA: una relación impredecible
7 abr 2022
La hélice α va antes que la hoja β y no es una espiral
También en 1951, Pauling y Corey publicaron la estructura de la hoja β, que recibió el apelativo de 'hoja' o 'lámina' (pleated sheet) por su aspecto plano en las proteínas fibrosas que habían analizado, y β porque se describió tras la α. Eran tiempos lógicos para la nomenclatura. En 1965 todos los implicados quedaron sorprendidos cuando se describió en las proteínas globulares que esas hojas (sheets) no formaban láminas plisadas (pleated) sino que estaban retorcidas (twisted).
1 abr 2022
Cosmos, astros, con sus ciencias y pseudociencias
No es lo mismo astronomía que astrología. Pero ¿qué pasa con cosmogonía y cosmología? ¿Son tan sinónimos como universo y cosmos? ¿Hay sinonimia, polisemia y homonimia entre ellos? Vamos por partes, como las integrales.
Los griegos llamaban κόσμος (kósmos) al universo y de ahí que se consideren sinónimos. De cosmos derivan dos cosas muy distintas: cosmogonía y cosmología. La cosmogonía es el conjunto de principios y modelos sobre el origen del universo visto desde el punto de vista filosófico y epistemológico. Está presente en todas las religiones para explicar el origen del universo ligado a elemento mitológicos. En cambio, la cosmología se acuñó en el siglo XVIII para referirse a la concepción global del universo que hoy se aplica a la parte de la astronomía que se ocupa de la dinámica, las estructuras y los modelos sobre el origen del universo. Por último, está el cosmismo, una corriente filosófica rusa iniciada con la última obra de Nikolái F. Fiódorov publicada en 1906 (poco después de su muerte) que trata de explicar la vida, pero también la misión que el hombre tiene para transformarla y perfeccionarla. Se la considera precursora del transhumanismo actual.
- Debemos dejar la astrología, la cosmogonía y casi que también el cosmismo fuera de las típicas ciencias.
- La astronomía se resume a la observación de los cuerpos celestes, y abarca tanto la astrofísica (para la mecánica de los componentes del universo) como la cosmología (para el estudio y la evolución del universo como un todo).
27 ene 2022
Células madre hay más de una

Las células madre (stem cells), células troncales o citoblastos tienen la capacidad de dividirse («proliferación celular») asimétricamente, dado que una célula hija será como la progenitora (autorrenovación) y la otra se diferenciará en una célula especializada. Tienen distinta potencia celular (cell potency, esto es, la capacidad para generar distintos tipos de células), que está en función de la parte del genoma que permanece sellada (genomic imprinting, que no se debe confundir con el silenciamiento génico o gene silencing) normalmente por superenrollamiento.
Si atendemos a su potencia, se dividen en:
- Totipotentes (totipotent): capaces de formar un organismo entero, desde las tres capas embrionarias y el tejido reproductor a los tejidos extraembrionarios (como la placenta). Ejemplos claros: una espora o un cigoto. Son capaces de autorrenovarse y proliferar sin problemas, y se puede controlar la expresión de todo el genoma.
- Pluripotentes (pluripotent): como las totipotentes, salvo que no consigue formar tejidos extraembrionarios ni, por tanto, el individuo entero. En ellas y las demás células madre, cada vez hay más genoma sellado que no se puede reactivar, lo que limita los tipos celulares que puede regenerar.
- Multipotentes (multipotent): tienen menos capacidad de proliferar y autorrenovarse que las anteriores (o sea, más genoma sellado), aunque la suficiente para diferenciarse en los tipos celulares de su misma capa o linaje embrionarios (epidermis, sistema nervioso y otros procedentes del ectodermo; tejido conjuntivo, muscular u otros procedentes del mesodermo; o los epitelios procedentes del endodermo). Se denominan también tissue-specific stem cells (citoblastos histoespecíficos), así como células progenitoras (progenitor cell) o incluso todo junto: células madre progenitoras multipotentes. Entre las más conocidas están las células madre hematopoyéticas o hemocitoblastos.
- Oligopotentes (oligopotent): solo son capaces de diferenciarse en algunos de los tipos celulares de su mismo linaje, o sea, una especie de multipotentes con limitaciones. Por ejemplo, los mielocitoblastos (myeloid stem cell) y los mielohemocitoblastos (bone marrow stem cells) se diferencian en menos tipos celulares que los hemocitoblastos (que son multipotentes).
- Unipotentes (unipotent): únicamente son capaces de diferenciarse en un solo tipo de tejido. No está claro si existen, porque muchas son realmente bipotentes. En la práctica, es sinónimo de hemoblasto (blast cell) y célula precursora (precursor cell), pero no de
célula progenitora, como hemos visto más arriba.
Para muchos autores, son las células multipotentes o progenitoras las que se subdividen en oligopotentes y unipotentes, pero esto es una minucia comparado con el enorme interés que suscitan en la medicina regenarativa.
- Embrionarias: proceden de la masa celular interna del blastocisto (una fase muy temprana del desarrollo embrionario), por lo que originarán las tres capas germinales; se han logrado mantener en cultivo.
- Perinatales: las que se han encontrado en el líquido amniótico y en la sangre del cordón umbilical; muy útiles, aunque es una lástima que no se puedan usar por la enorme controversia bioética.
- Adultas: las que aparecen en la mayoría de los tejidos de un organismo adulto que se renuevan o regeneran periódicamente cuando acaece algún daño tisular. Aunque prometedoras para la investigación, parecen no ser tan versátiles y duraderas como las embrionarias, además de tener mayor propensión a contener alteraciones genéticas.
- Inducidas o iPS (del inglés induced pluripotent stem cells): gracias a la reprogramación genética artificial de una célula somática diferenciada se ha conseguido generar células madre pluripotentes con su mismo potencial de crecimiento y diferenciación. Por tanto, son tan versátiles como las embrionarias y perinatales, pero sin su controversia. El diseñador de esta técnica, el japonés Shinya Yamanaka, recibió por ello el Nobel en 2012.
14 jul 2021
Sinónimos cancerosos
Neoplasia (neoplasm, neoplasia), término que hemos importado del francés (néoplasie) que significa «tejido de nueva formación», se utiliza para designar a toda esta familia de enfermedades. Las células de este tipo son neoplásicas (neoplastic). Las hay benignas y malignas, pero cuando va sin adjetivo, casi siempre hace referencia a las malignas.
Tumor (tumour [UK], tumor [US]), que originalmente hacía referencia a cualquier bulto o hinchazón (tumefacción, tumoración), desde el siglo XX solo se emplea para una masa sólida de células. Cuando se trate de una masa benigna (tumor benigno), sería sinónimo de neoplasia benigna. Pero el tumor maligno no es sinónimo estricto de neoplasia maligna dado que, por ejemplo, una leucemia no es un tumor al no presentar masas sólidas. Las células de estas masas, benignas o malignas, son tumorales.
Carcinoma (carcinoma, carcinomata), del griego καρκίνωμα (karkínōma), hace referencia a las neoplasias malignas que se forman en los tejidos epiteliales (la gran mayoría). Sus células son carcinomatosas.
Sarcoma (sarcoma, sarcomata) es un término que deriva del griego σάρκωμα (sárkōma → crecimiento de la carne, tumor carnoso). Son las neoplasias malignas que se forman en un tejido conjuntivo (incluido el músculo que forma la carne), como el sarcoma de Rous. Las células de este tipo son sarcomatosas.
Cáncer (cancer), una palabra de origen latino (que a su ver procedía del término griego para «cangrejo») que acuñó Hipócrates, se usa cada vez más como sinónimo de neoplasia maligna (como se señalan Navarro y la Wikipedia). Al principio solo hacía referencia a las neoplasias epiteliales y era sinónimo de carcinoma, pero hoy el carcinoma es un tipo de cáncer. Este tipo de células son cancerosas (cancerous).
Teratoma (teratoma) que hemos tomado del latín y que procedía del griego τέρας, -ατος (téras, -atos → 'monstruo') y -oma → 'hinchazón', es un tumor de origen embrionario que se suele detectar en la edad adulta en forma de quiste. Muchos son benignos, pero los inmaduros sí que podrían ser malignos. Se consideran sinónimos cuyo uso se desaconseja disembrioma (dysembryoma), teratoblastoma, y tumor teratoide (teratoid).
Zaratán: derivado del árabe con el significado de cangrejo, empezó siendo sinónimo de cáncer y de carcinoma, para acabar haciendo referencia solo al cáncer de mama desde al menos 1739. Sin embargo, es muy poco probable que alguien utilice este precioso vocablo en nuestros días.
15 jun 2021
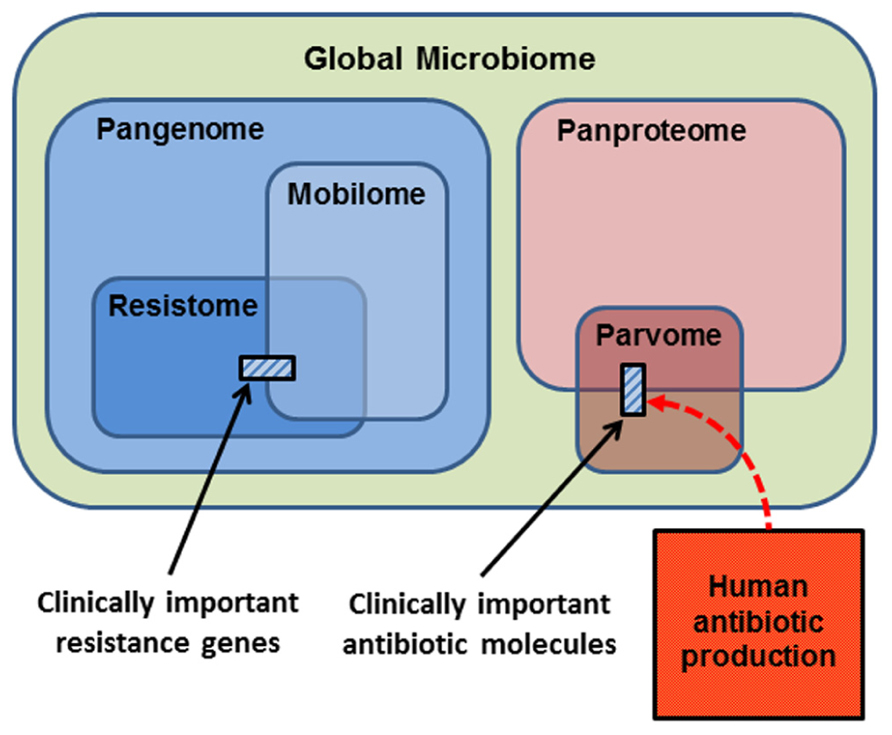
¿Mobiloma o moviloma?
Hace unos meses vimos que, por influencia del término 'genoma', resulta muy habitual que se añada el sufijo -oma para indicar un conjunto de elementos que conforman una entidad de interés biológico, como epigenoma, metagenoma, transcriptoma, proteoma, interactoma, viroma, etc.
Añadamos ahora otro término que no debería suscitar dudas a un traductor, pero que los científicos no tienen tan claro. Me estoy refiriendo a mobilome, definido como el conjunto de elementos genéticos capaces de desplazarse dentro de un genoma y entre genomas. Con frecuencia, se considera que un DNA móvil es sinónimo de transposón, así que podríamos haberlo denominado transposoma. Pero esto tiene un problema: los elementos móviles (MGE: mobile genetic elements) del mobilome incluyen los transposones, y también plásmidos, virus y otros parásitos del genoma (principalmente intrones autoayustables e inteínas). Así que, para abarcar todos estos MGE, debemos usar moviloma. No tiene ningún sentido calcar el uso de la 'b' que aparece en la Wikipedia y en otras páginas por las mismas razones por las que nuestros teléfonos son móviles (y no móbiles, lo que haría referencia a muebles e inmuebles, que nada tienen que ver con la movilidad).
Quiero agradecer a @SilvanaTapia3, compañera de la UMA, su aporte para esta entrada.
2 jun 2021
Y el genoma más grande es…
En la entrada anterior quedó colgada la pregunta de si los humanos tenemos el genoma más grande del planeta Tierra. La respuesta breve es no. Veamos los motivos.
En 2021 se acaba de publicar el genoma animal secuenciado más grande hasta ahora: el del pez pulmonado Neoceratodus forsteri , cuyo interés reside en ser considerado un fósil viviente de cómo los vertegrados acuáticos conquistaron el medio terrestre. El bicho tiene nada menos que 34,5 Gpb (al principio se creyó que llegaba a las 43 Gpb), con 31 120 genes, una cantidad de DNA repetitivo superior al 90 % (en los humanos es del 55%), y cuyos intrones son de media 10 veces más largos (50 kpb) que en los humanos (6 kpb). Por tanto, tiene ~10× más genoma, ~1,5× más genes con intrones ~8 veces mayores y casi el doble de DNA repetitivo. No es el único, pues hay otro pez pulmonado, Protopterus annectens, que tiene un genoma igual de inmenso, en torno a 40 Gpb. O sea, que ya se han secuenciado genomas unas 10 veces mayores que el humano.Pero no acaba aquí la cosa, porque las plantas también nos pueden hacer enrojecer de vergüenza. Al tener un metabolismo secundario, la mayoría de los vegetales poseen más genes que un animal. Pero es que el genoma del pino y otras gimnospermas tiene 22 Gpb (7× el humano). La planta con el genoma más grande conocido hasta ahora (150 Gpb), aunque no secuenciado, es Paris japonica, 50× mayor que el humano.
¿Hay algo todavía más grande? Pues sí, y con esto apuñalamos del todo lo que queda de nuestro ego de ser superior: el genoma de dos amebas. Por un lado está Amoeba proteus, con 290 Gpb repartidas en 500 cromosomas, y por otro su congénere Polychaos dubium (antes conocido como Amoeba dubia) con nada menos que 670 Gpb (el más grande conocido hasta ahora). Estos megagenomas se describieron en 1968, aunque en 2004 se sugirió que quizá habría que replantearse los cálculos a la baja.
21 may 2021
¿Es grande el genoma humano?
En otra entrada vimos el éxito de 'genoma' desde el punto de vista lingüístico. Ahora vamos a abordar otro éxito completamente diferente.
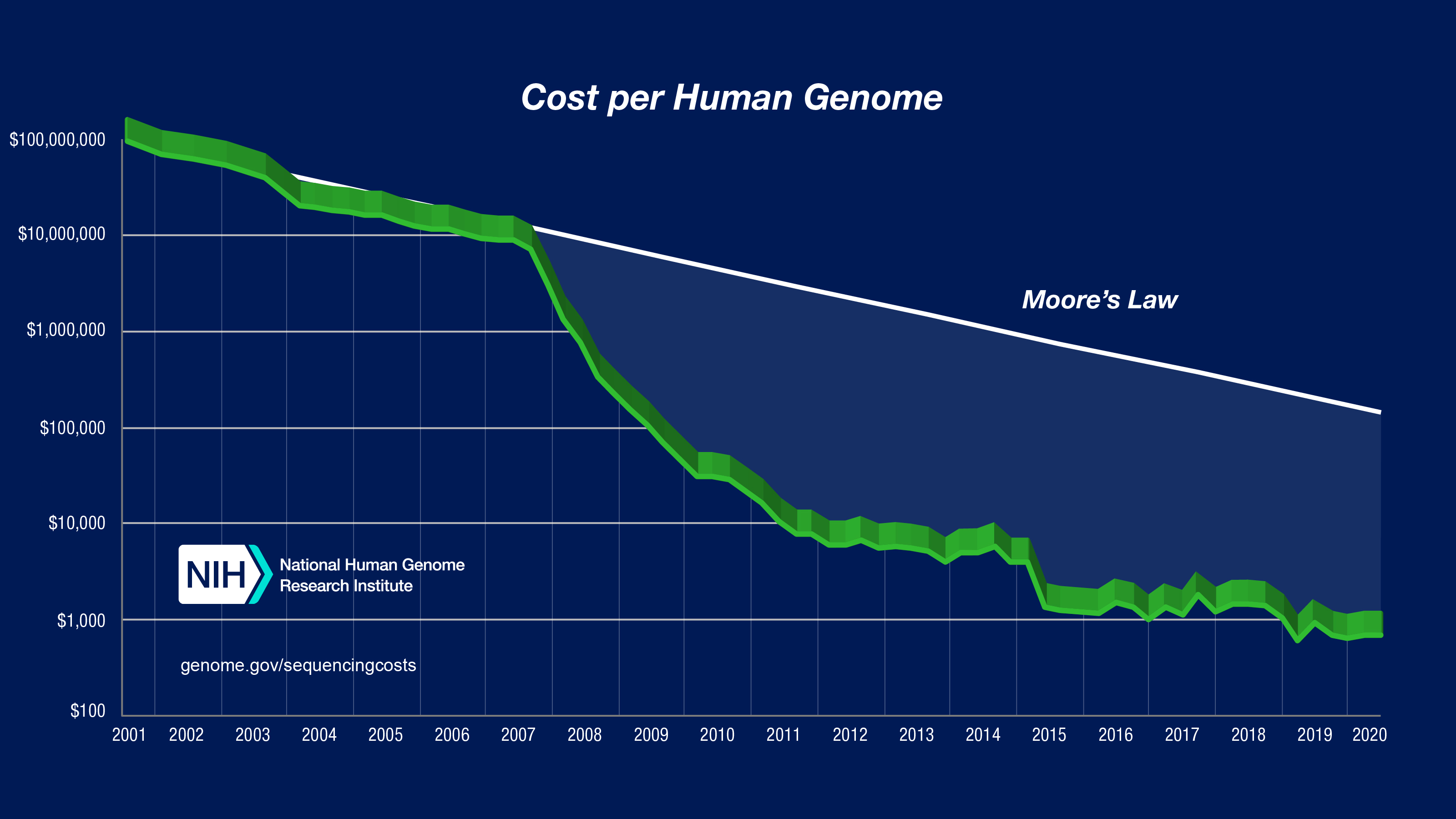
Cuando en 1984 se empezaron a plantear si sería posible secuenciar el genoma humano, el grupo de Craig Venter ya estaba secuenciando el primer procariota (Hemophilus imfluenzae) con el que dio a conocer el sistema de secuenciación indiscriminada (shotgun sequencing), y en Europa estábamos (sí, yo puse mi grano de arena) poniendo en marcha la secuenciación del primer eucariota (la levadura Saccharomyces cerevisiae). El debate estaba en que abordar la secuenciación de nuestro genoma iba a tener un coste excesivo dadas las pocas secuencias codificantes que contenía (que se creía, inocentemente, que constituían el 10%; si llegan a saber que solo era un 1,1%, igual ni empiezan). En la imagen adjunta se puede ver lo que costó el primer genoma humano y cómo ha ido decayendo el coste con los años y los avances tecnológicos.
Tras poner en canción a un montón de grupos e investigadores, en 2001 se publicó el primer borrador y se indicó que era 25 veces más grande que cualquier otro conocido por entonces. En 2003 se publicó la secuencia de la eucromatina humana (el 92,1%). Hoy, 20 años después del primer hito, sabemos que contiene 20 437 genes que codifican proteínas (coding genes) y otros 23 988 genes no codificantes (non-coding genes), en un genoma de 3,096 Gpb que produce más de 235.000 transcritos diferentes.
Se han secuenciado cientos de genomas, desde virus a eucariotas complejos, y se ha visto que el de muchos animales y plantas oscila entre 0,4 y 0,7 Gpb (un gigapar de bases corresponde a 1 millón de pares de bases). Por tanto, nuestro genoma ya no es más que unas 10 veces mayor que el de muchos otros seres vivos. Así que de 25× en 2001 hemos pasado a un escaso 10× en 2021. O lo que es más denigrante para tantísimos homocéntricos: nuestro genoma es solo ligeramente superior al del ratón (2,72 Gpb) y tiene menos genes codificantes que una mala hierba, el arroz y, en general, que cualquier planta. Así que ¿tendremos el genoma más grande del planeta? La respuesta llegará en la próxima nanoentrada.
6 may 2021
Proteínas metamórficas, camaleónicas, o mejor metamorfas
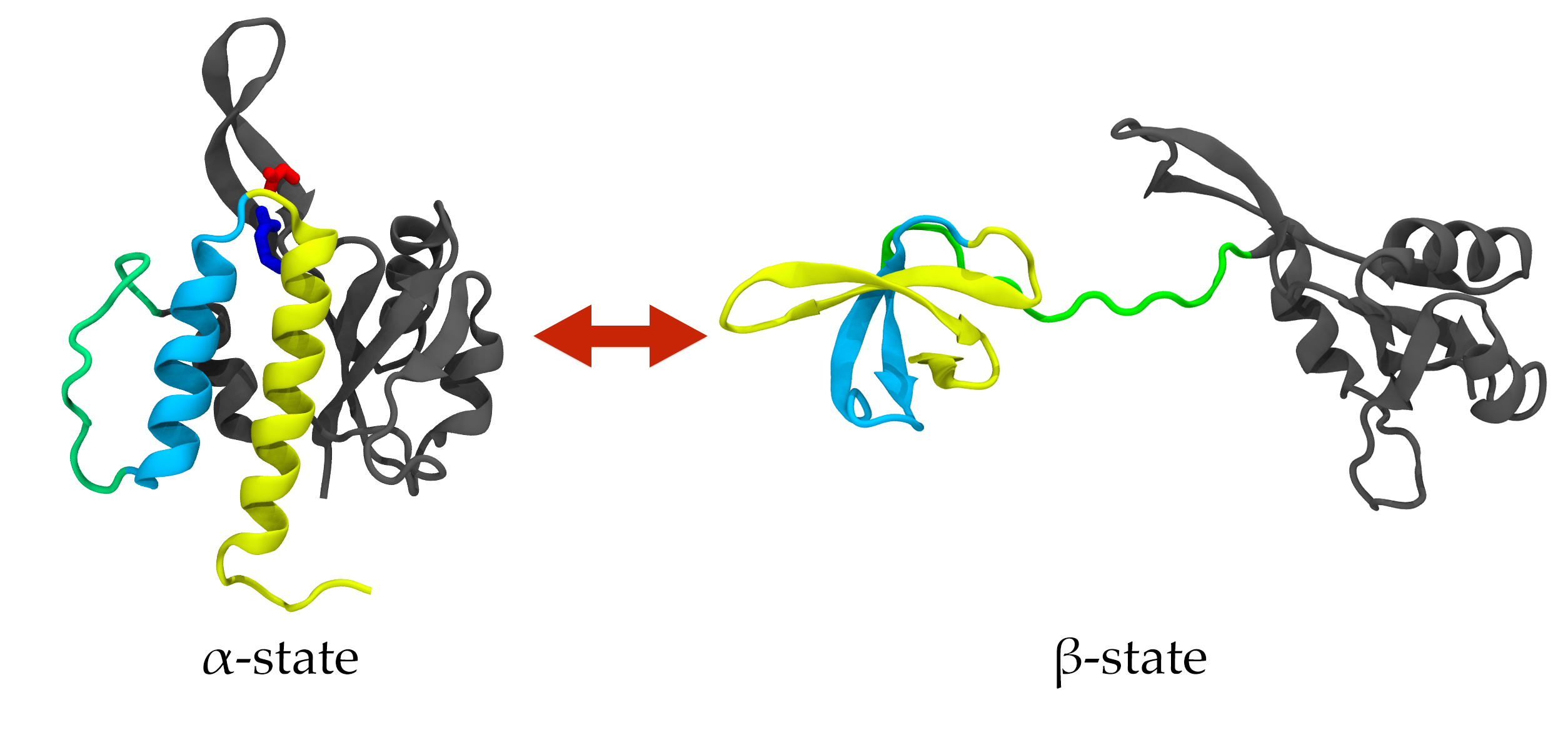
¿Son las proteínas metamórficas producto del metamorfismo, como las rocas que llevan el mismo adjetivo? Pues no, no son más que el producto de una traducción calquista del inglés metamorphic proteins, para variar, porque realmente derivan de metamorfosis, que tampoco es exclusiva de los insectos. Y es que el término metamórfico solo tiene asignado un significado en el ámbito de la geología en el DLE y seguramente en la mente de muchos de nosotros. Por eso, hubiera sido mucho más sensato acuñar y usar metamorfo, puesto que contamos en nuestro idioma con el sufijo -morfo para referirnos a la forma, como actinomorfo, alomorfo, zigomorfo, etc. Contamos también con otros términos más afines a este problema, como amorfo, dimorfo y polimorfo, por lo que metamorfo hubiera sido muy coherente. Por desgracia, se calca «metamófico» como adjetivo de «metamorfosis» en los contextos no geológicos, y me estoy temiendo que va a ocurrir lo mismo que con «hidrófobo», con el que la presión del inglés ha hecho que la RAE acepte el calco hidrofóbico. Ya falta poco para hibridizarnos del todo.
Una vez terminadas las disquisiciones lingüísticas, ¿qué son esas proteínas metamorfas (o metamóficas)? La teoría del plegamiento de las proteínas (protein folding) ya quedó establecida en 1973 por Anfinsen, con la idea de que una proteína solo adquiere una estructura tridimensional funcional. Pero 10 años después ya se empezaban a encontrar secuencias de aminoácidos que eran capaces de alternar entre dos plegamientos que le daban dos funciones distintas, como la RfaH de la imagen. En 1996 se reunieron bajo la denominación secuencias camaleónicas (chameleon sequences) todas las cadenas de aminoácidos capaces de adoptar más de una estructura de manera intercambiable (no como la β-amiloide del alzhéimer o la Prp de los priones, que cambian irreversiblemente). Con frecuencia, secuencia camaleónica, proteína metamórfica, y el híbrido proteína camaleónica se usan como si fueran sinónimos. Pero, como hemos indicado, el primero (secuencia camaleónica) hace referencia a la secuencia de aminoácidos, mientras que «proteína metamorfa» debería reservarse para la estructura adquirida, porque una secuencia camaleónica producirá al menos dos proteínas metamorfas. Seguimos sin tener claro por qué existen este tipo de proteínas, que inicialmente se creía que eran una etapa intermedia de evolución del plegamiento ya que se creía que cada proteína se plegaba de una sola forma. En 2023, ya se empieza a pensar que se trata de una capacidad adaptativa que se ha conservado y optimizado a lo largo de la evolución.
Quiero agradecer a los compañeros de @Tremedica el debate que me ayudó a conformar esta entrada.